Rigspolitiet indstiller til at øge antal STR-markører for DNA-beviser til 16
Fornylig har danske medier bragt historien om at Rigspolitiet indstiller til at antal DNA-markører for DNA-beviser øges fra 10 STR-markører til 16 STR-markører. Forholdet mellem sandsynlighederne for sammenfald mellem to DNA-profiler falder med mere end en faktor 500,000,000.
Rigspolitiet: Opdatering af DNA profiler
Uddrag fra Rigspolitiets skrivelse:
Rigspolitiet har efter drøftelse med Rigsadvokaten den 21.10 2019 besluttet at skærpe retningslinjerne for opdatering af DNA-referenceprofiler, der er typebestemt efter 10 DNA-systemer. Denne typebestemmelse blev anvendt frem til 2012. Fra 2012 er der anvendt typebestemmelse efter 16 DNA-systemer.
DNA profiler
DNA betragtes med rette som et af de stærkeste beviser i opklaring af kriminalsager. I modsætning til fx fingeraftryk, giver forståelsen af den biologiske overlevering fra generation til generation mulighed for at modellere hyppighederne af en given DNA-profil, hvilket bruges for at kunne vurdere den bevismæssige vægt af et DNA-spor (Link til Advokatsamfundets tekst om DNA-beviser)
Når man taler om DNA-profiler til personidentifikation, som vil være tilfældet hvor man ønsker at undersøge om en given mistænkt kan knyttes til et gerningssted, benyttes oftest såkaldte short tandem repeat-markører der forkortes til STR-markører. STR-markører er veldefinerede steder på en persons DNA hvor der i populationen er observeret et tilstrækkelig stor variation individerne imellem. Dette gør at sandsynligheden for overlappende DNA-profiler er lille, hvilket igen gør at sammenfald mellem en mistænkts DNA profil og profilen fra et gerningssted er inkriminerende for den mistænkte.
DNA nedarves fra generation til generation ved at ét af morens kromosomer og ét af farens kromosomer giver anledning til barnets kromosompar (når vi ser bort fra rekombinations hændelser). Vi mennester har i alt 23 kromosompar, hvoraf de ene er kønskromosomerne (XX for kvinder og XY for mænd). På de resterende autosomale kromosomer kan man ved kun at betragte barnets DNA profil (også kaldet genotype) ikke afgøre hvilket kromosom som nedstammer fra moren og hvilket der nedstammer faren. En STR-markør kan antage et variabelt antal tilstande som kaldes alleler, hvor altså der arves én allel fra moren og én fra faren. Den typiske teknologi som benyttes til at analysere en DNA-profil angiver allelerne som heltal, fx (7,9) hvilket betyder at personen har arvet hhv allele 7 og 9 fra sine forældre. Tallele refererer til hvor mange gange en given DNA-sekvens er gentaget. Ofte er den gentagne sekvens af længde fire, fx ACAT hvor bogstaverne henviser til de fire mulige DNA-baser. Et allel 7 er derfor angivet ved ACATACATACATACATACATACATACAT. Vi kalder profiler med to forskellige alleler for heterozygote og homozygote hvis de nedarvede alleler fra forældrene er ens.
Indtil 2012 benyttede man i Danmark 10 STR-markører således at man havde 10 par af alleler. STR-markørerne er udvalgt til være statistisk uafhængige af hinanden. Ligeledes antager man ofte også uafhængig mellem allelerne inden for en given STR-markør. Det betyder at sandsynligheden for at observerer genotypen hvor
er allele frekvensen (hyppigheden) af allel 7 (tilsvarende for allele 9) og hvor faktoren på 2 skyldes at (7,9) kan fremkomme på to måder (idet vi ikke kan se hvilket allel der kommer fra hvilken forældre). Uafhængigheden mellem STR markørerne gør at vi kan gange bidragene fra de forskellige STR-markører sammen hvorved vi slutteligt opnår ganske små sandsynligheder for den specifikke DNA-profil, fx i størrelsesordenen
.
Usandsynlige hændelser?
Allerede i 2001 blev man i USA bekymret over at antageligt urelaterede personer havde nær sammenfald af DNA-profiler. Med nært sammenfald menes der at man enten matcher på begge alleler på en STR-markør eller matcher på ét af de to alleler. Har vi fx DNA-profilerne og
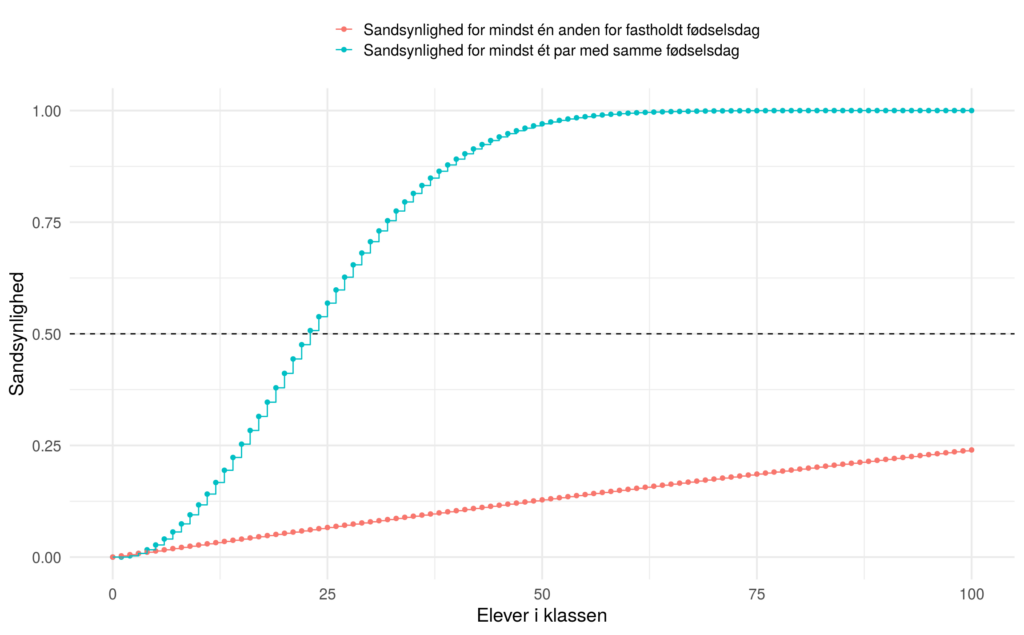
vil de matche på én markør (den sidste hvor begge har alleler 13 og 14), delvist matche på den anden markør (overlapper på allel 11) og mismatch på den første markør (ingen alleler tilfælles). Weir (2004, 2007) beskrev hvorfor sådanne sammenfald ikke er overraskende selvom hyppighederne for DNA-profiler er ganske små. Han sammenlignende fænomenet med det såkaldte “fødselsdagsproblem”, som mange har stiftet bekendtskab med i deres introduktion til sandsynlighedsregning: Hvor mange elever skal man have i en klasse før sandsynsynligheden for mindst ét par har samme fødselsdag overstiger 50%?. Som det ses nedenfor (blå kurve) har man for 23 elever mere end 50% chance for to personer med samme fødselsdag. Det kan virke intuitivt forkert når nu året har 365 dage – men svaret er at vi ikke har angivet hvilken dato der er tale om. Fastholder vi datoen får vi den røde linje, hvilket er sandsynligheden at mindst èn anden har samme fødselsdag som fx dig. For den blå kurve laver vi alle parvise sammenligninger for eleverne, hvilket svarer til
sammenligninger som for
svarer til 253 sammenligninger.
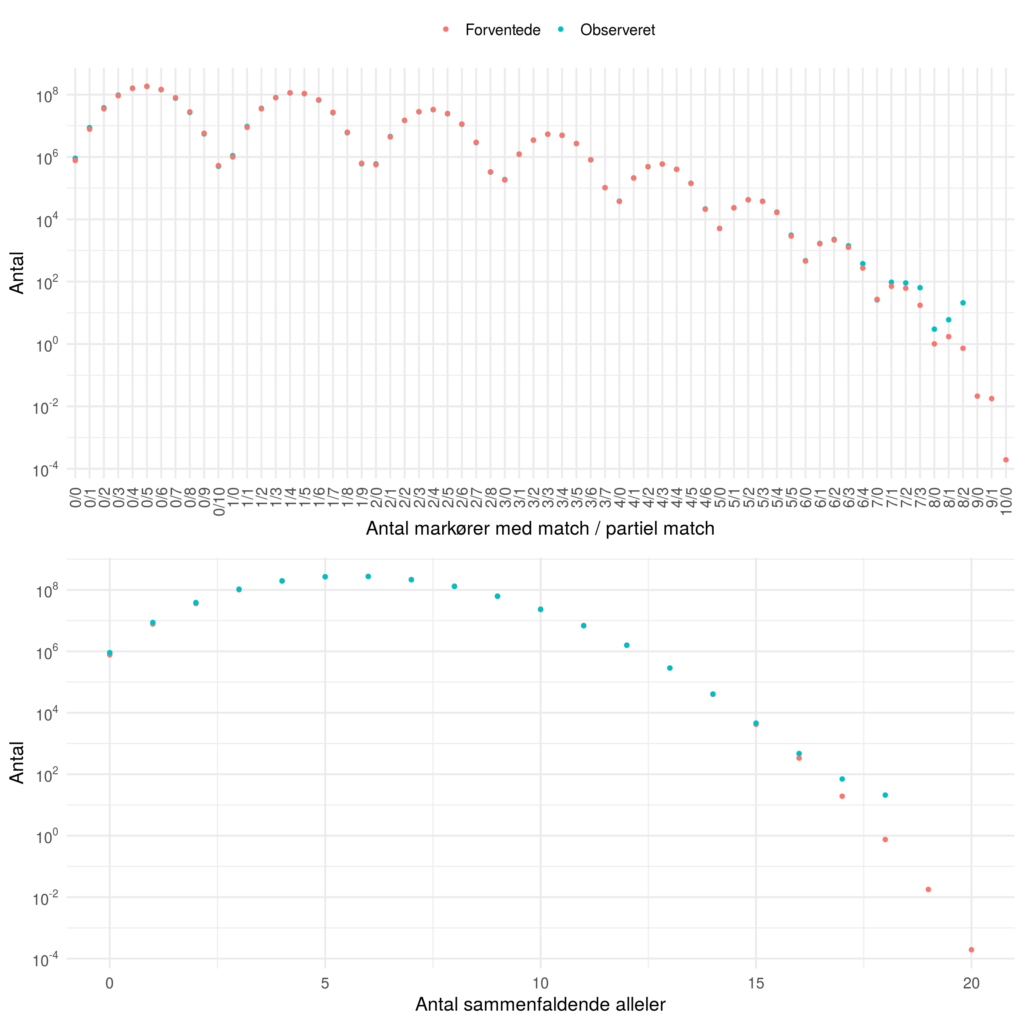
Det samme gør sig gældende for DNA profiler i en database. Hvis vi undersøger sammenfaldet mellem hvert par af DNA-profiler bliver det lynhurtigt til rigtigt mange sammenligninger. I 2012 udgav vi en artikel hvor vi sammenlignede 51,517 danske DNA profiler som var genotypet på 10 STR-markører. For et par af DNA-profiler udledte Weir at for en enkelt markør er sandsynlighederne for forskellige grader af overlap givet ved:
,
,
hvor , er summen af allel frekvenser opløftet i
’te.
Vi fandt efterfølgende en effektiv måde at beregne det forventede antal DNA profiler med markører med fuldt match og
markører med delvist match (partial match). Dette kan også omregnes til at DNA profilerne har
alleler tilfælles. For de 51,517 DNA-profiler fandt vi at teorien og praksis stemte glimrende overens (afgivelserne for høje antal sammenfaldende alleler kan forklares af øget usikkerhed på små sandsynligheder samt der kan korrigeres for sub-populations effekter – det er undladt her.)
Foruden rene DNA-profiler optræder DNA-profiler ofte i blandinger af to eller flere DNA-profiler, hvilket kaldes DNA-miksturer eller DNA-blandinger. Det betyder at man ikke entydigt kan fastslå bidragsyderne til en given blanding, hvilket øger antallet af mulige DNA-profiler som skal sammenlignes. Risikoen for sammenfald mellem DNA-profiler vokser altså når man også tager DNA-blandinger i betragtning.
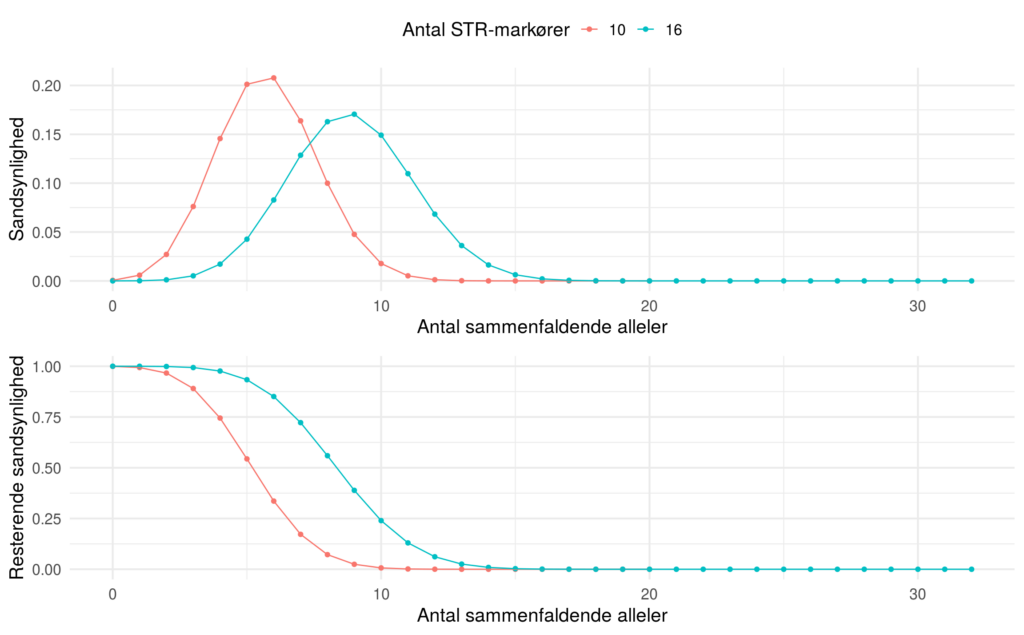
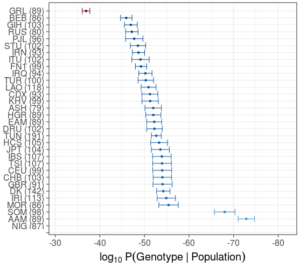
Effekten af at øge antal markører fra 10 til 16 STR-markører
I Rigspolitiets skrivelse indstilles der til at øge antallet af STR-markører fra 10 STR-markører til 16 STR-markører. Nedenfor ses hvordan sandsynlighederne påvirkes af at øge antal STR-markører på ovenstående udtryk. Risikoen for nær sammenfald af DNA-profiler mindskes altså betragteligt når antallet af mulige fælles alleler vokser til 32 fra 20. Faktisk er forholdet mellem og
givet ved 545,969,753 – altså en faktor på mere end 500 mio.
Referencer
B.S. Weir (2007) The rarity of DNA profiles, Annals of Applied Statistics 1 (2): 358–370.